|
|
自我网络 Ego Networks
导入数据
在本节中,我们从 2004年的GSS网络模块数据中 分析 自我网络。我们将使用 GSS数据 来熟悉网络密度和异质性的度量方法。它还将教会我们如何同时分析多个网络。在某些情况下,您可能有数百个完整的网络——例如,关于高中的数据通常有来自许多不同高中的网络。由于学校是分开的,你必须分别分析它们,但一个一个这样做是费力的。在这里,我们将学习自我网络以及将同样的功能应用到许多网络的策略:
library(igraph)
gss <- read.csv(&#34;https://raw.githubusercontent.com/mahoffman/stanford_networks/main/data/gss_local_nets.csv&#34;,
stringsAsFactors = TRUE) 让我们看看数据。你可以在你的环境变量中找到并且点击它,或者在命令框中直接键入View(gss)
> head(gss)
sex race age partyid relig numgiven close12
1 female other 52 independent catholic 0 NA
2 female other 43 not str republican catholic 0 NA
3 male black 52 strong democrat protestant 4 1
4 female other 34 ind,near dem catholic 4 2
5 male other 22 ind,near dem moslem/islam 0 NA
6 male black 26 not str democrat protestant 6 0
close13 close14 close15 close23 close24 close25 close34 close35 close45
1 NA NA NA NA NA NA NA NA NA
2 NA NA NA NA NA NA NA NA NA
3 2 0 NA 2 2 NA 1 NA NA
4 0 2 NA 2 2 NA 2 NA NA
5 NA NA NA NA NA NA NA NA NA
6 2 1 1 1 1 1 2 2 2
sex1 sex2 sex3 sex4 sex5 race1 race2 race3 race4 race5 educ1 educ2
1 NA NA NA NA NA NA NA NA NA NA NA <NA>
2 NA NA NA NA NA NA NA NA NA NA NA <NA>
3 1 1 0 0 NA 1 1 1 1 NA 1 h.s. grad
4 1 0 1 1 NA 2 2 2 2 NA 1 h.s. grad
5 NA NA NA NA NA NA NA NA NA NA NA <NA>
6 1 1 0 1 1 0 1 1 2 2 1 h.s. grad
educ3 educ4 educ5 age1 age2 age3 age4 age5 relig1
1 <NA> <NA> <NA> NA NA NA NA NA <NA>
2 <NA> <NA> <NA> NA NA NA NA NA <NA>
3 Grad Bachelors <NA> 56 40 58 59 NA protestant
4 Grad Grad <NA> 63 36 34 36 NA catholic
5 <NA> <NA> <NA> NA NA NA NA NA <NA>
6 h.s. grad Some College Some College 25 25 39 33 30 other
relig2 relig3 relig4 relig5
1 <NA> <NA> <NA> <NA>
2 <NA> <NA> <NA> <NA>
3 protestant protestant protestant <NA>
4 catholic catholic catholic <NA>
5 <NA> <NA> <NA> <NA>
6 other catholic catholic catholic一共有42个变量。前五个与特定受访者的属性有关:他们的性别、年龄、种族、党派和宗教。
接下来的36个组成了GSS网络模块的“网络”部分。这种结构可能有点令人困惑,特别是如果你没有读过任何使用这种数据的论文。该模块的基本想法是询问人们在过去6个月里与他们讨论过“重要问题”的最多5个人的情况。受访者报告了他们讨论“重要问题”的人数:这是我们数据集中的变量“numgiven”。他们还被要求详细描述这五个人之间的关系:他们是特别亲密,彼此认识,还是完全陌生。这与数据集中的接近变量一致,例如,close12是每个应答者1与2的接近度。最后,他们被问及自我网络中最多五个人的属性(性别、种族、年龄)。
为了了解为什么这些被称为自我网络,让我们找一个被调查者,把他们自称讨论过“重要问题”的最多五个人的关系画出来。为此,我们必须首先将变量close12到close45转换为一个边列表,每个响应器对应一个边列表。
这需要一些复杂的代码。首先,我们使用grepl提取所需的列。Grep基本上使用字符串匹配,因此它会遍历列名并识别其中包含“close”这个词的列名(更多信息请查看这里:https://www.regular-expressions.info/rlanguage.html)
ties <- gss[,grepl(&#34;close&#34;, colnames(gss))]
head(ties)
> head(ties)
close12 close13 close14 close15 close23 close24 close25 close34 close35
1 NA NA NA NA NA NA NA NA NA
2 NA NA NA NA NA NA NA NA NA
3 1 2 0 NA 2 2 NA 1 NA
4 2 0 2 NA 2 2 NA 2 NA
5 NA NA NA NA NA NA NA NA NA
6 0 2 1 1 1 1 1 2 2
close45
1 NA
2 NA
3 NA
4 NA
5 NA
6 2形成网络矩阵
在该案例中,让我们首先制作一个矩阵,我们可以用给定应答者的亲密度值填充它。
mat = matrix(nrow = 5, ncol = 5)事实证明,我们可以把一个人的接近值直接分配到矩阵的下三角形。这里我们为受访者3。即只选取了ties数据框的第三行
mat[lower.tri(mat)] <- as.numeric(ties[3,])我们可以使矩阵对称,因为这里的关系(紧密度)是相互的(即关系是无向的)。
mat[upper.tri(mat)] = t(mat)[upper.tri(mat)]
mat
> mat
[,1] [,2] [,3] [,4] [,5]
[1,] NA 1 2 0 NA
[2,] 1 NA 2 2 NA
[3,] 2 2 NA 1 NA
[4,] 0 2 1 NA NA
[5,] NA NA NA NA NA现在让我们去掉任何一个缺失的应答者。即NA
na_vals <- is.na(mat)
non_missing_rows <- rowSums(na_vals) < nrow(mat)
mat <- mat[non_missing_rows,non_missing_rows]
> mat
[,1] [,2] [,3] [,4]
[1,] NA 1 2 0
[2,] 1 NA 2 2
[3,] 2 2 NA 1
[4,] 0 2 1 NA并将对角线设为零,因为NAs会给引语带来麻烦
> diag(mat) <- 0
> mat
[,1] [,2] [,3] [,4]
[1,] 0 1 2 0
[2,] 1 0 2 2
[3,] 2 2 0 1
[4,] 0 2 1 0我们可以使用这个矩阵为单个受访者创建一个网络,就像我们在上一篇教程中所做的那样,但这次使用的不是图表而是邻接函数,因为我们的输入数据是矩阵。我们将指定它是无向和加权的。
ego_net <- graph.adjacency(mat, mode = &#34;undirected&#34;, weighted = T)
plot(ego_net, vertex.size = 30,
vertex.label.color = &#34;black&#34;,
vertex.label.cex = 1)
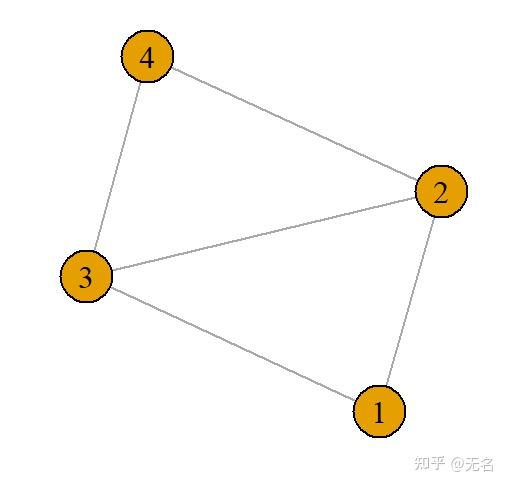
自我网络形成
唯一的问题是我们必须对数据集中的每一行都这样做,我们应该怎么做呢?一种选择是创建一个函数,该函数使用上面的代码将ties数据集中的任何行转换为自我网络,然后将该函数应用于数据中的每一行。下面就是这样一个函数!
make_ego_nets <- function(tie){
# 制作矩阵
mat = matrix(nrow = 5, ncol = 5)
# 将tie值分配给下面的三角形
mat[lower.tri(mat)] <- as.numeric(tie)
# 对称化
mat[upper.tri(mat)] = t(mat)[upper.tri(mat)]
# 识别缺失的值
na_vals <- is.na(mat)
# 确定所有值都缺失的行
non_missing_rows <- rowSums(na_vals) < nrow(mat)
# 查找每一行
if(sum(!non_missing_rows) > 0){
mat <- mat[non_missing_rows,non_missing_rows]
}
diag(mat) <- 0 #对角线为0
ego_net <- graph.adjacency(mat, mode = &#34;undirected&#34;, weighted = T)
return(ego_net)
}现在,我们可以使用lapply循环遍历数据中的所有行,并将上述函数应用于每一行。它将返回一个大小为nrow(ties)的列表,其中每一项都是数据中一个应答者的自我网。
ego_nets <- lapply(1:nrow(ties),
FUN = function(x) make_ego_nets(ties[x,]))
> head(ego_nets)
[[1]]
IGRAPH 0772b64 U--- 0 0 --
+ edges from 0772b64:
[[2]]
IGRAPH 0772e87 U--- 0 0 --
+ edges from 0772e87:
[[3]]
IGRAPH 0772f16 U-W- 4 5 --
+ attr: weight (e/n)
+ edges from 0772f16:
[1] 1--2 1--3 2--3 2--4 3--4
[[4]]
IGRAPH 0772f9c U-W- 4 5 --
+ attr: weight (e/n)
+ edges from 0772f9c:
[1] 1--2 1--4 2--3 2--4 3--4
[[5]]
IGRAPH 0773016 U--- 0 0 --
+ edges from 0773016:
[[6]]
IGRAPH 0773096 U-W- 5 9 --
+ attr: weight (e/n)
+ edges from 0773096:
[1] 1--3 1--4 1--5 2--3 2--4 2--5 3--4 3--5 4--5
现在我们有一长串的网络。让我们随便看看一个网络,比如说第1001个自我网络。
random_ego_net <- ego_nets[[1021]]
plot(random_ego_net)
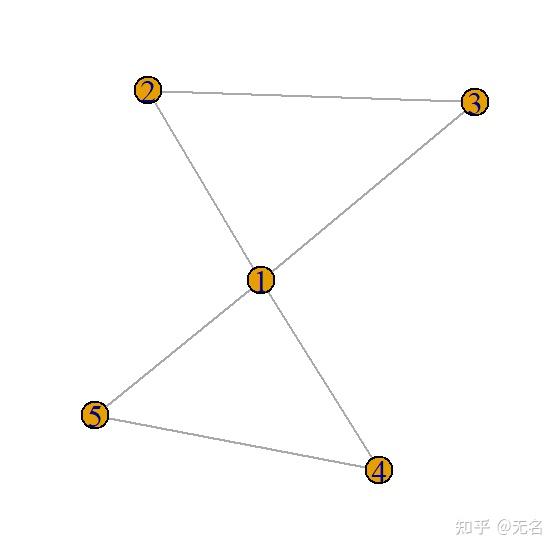
其中一个自我网络。
网络的大小和密度
现在我们有了一个网络列表,我们可以使用一行代码对每个网络应用相同的函数,同样是在lapply的帮助下。网络大小是指网络中节点的数量。为了找到它,我们使用vcount()函数。我们还可以使用ecount()来查找边的数量
network_sizes <- lapply(ego_nets, vcount)
network_edge_counts <- lapply(ego_nets, ecount)
head(network_sizes)
> head(network_sizes)
[[1]]
[1] 0
[[2]]
[1] 0
[[3]]
[1] 4
[[4]]
[1] 4
[[5]]
[1] 0
[[6]]
[1] 5我们可以通过简单地将列表转化为一个向量,并在结果向量上使用均值函数来求其中一个结果的均值。
network_sizes <- unlist(network_sizes)
mean(network_sizes, na.rm = T)
> mean(network_sizes, na.rm = T)
[1] 1.796634我们同样可以画出网络的直方图,查看网络数据的分布状况。
hist(network_sizes,
main = &#34;Histogram of Ego Network Sizes&#34;,
xlab = &#34;Network Size&#34;)
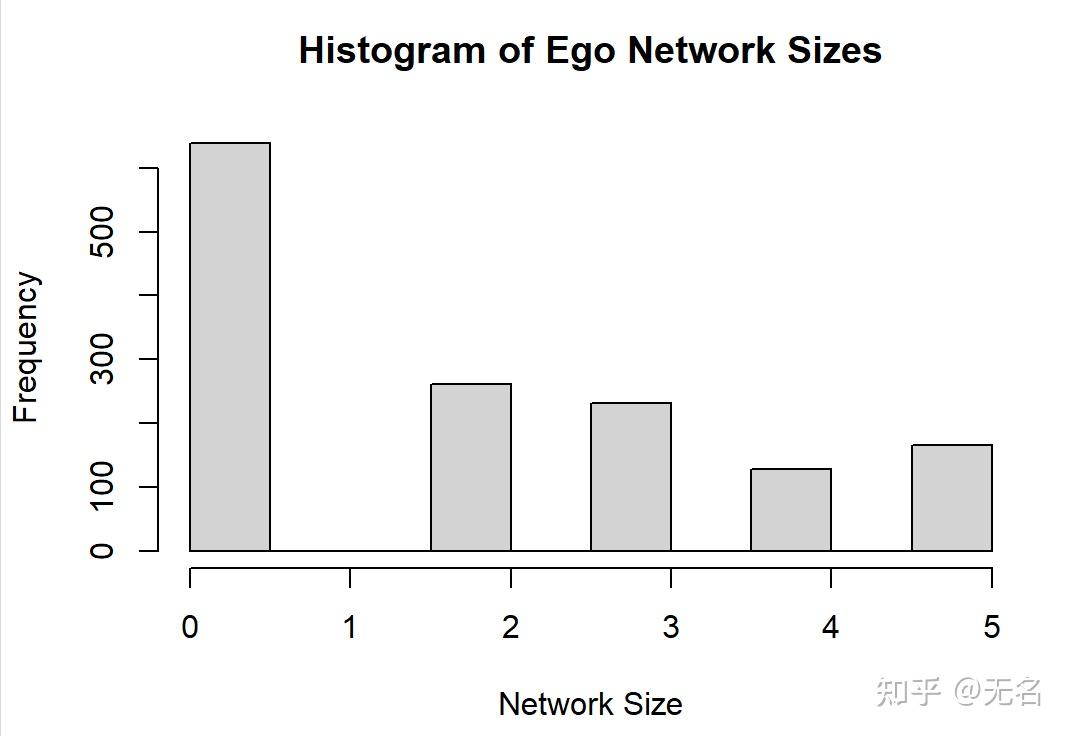
当然,我们也可以对 边 做同样的处理。
network_edge_counts <- unlist(network_edge_counts)
hist(network_edge_counts, main = &#34;Histogram of Ego Network Edge Counts&#34;, xlab = &#34;# of Edges&#34;)
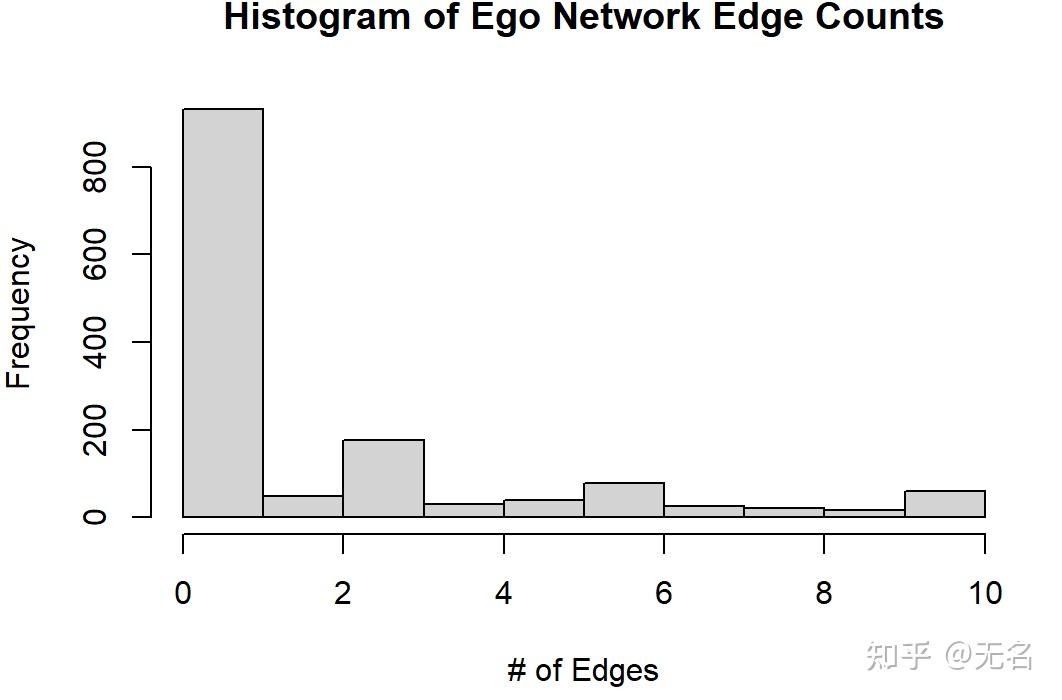
最后,让我们试试密度。密度表示网络中有多少条边,除以可能的总边数。在规模为N的无向网络中,可能有(N * (N-1))/2条边。如果你回想一下每个网络下面的矩阵,N * N-1指的是行数(应答者)乘以列数(又是应答者)减1,这样就排除了对角线(即与自己的关系)。在无向网络中,我们把这个数除以2只是为了说明网络是对称的。
对于之前笔记中的的随机自我网络,我们可以自己计算如下。
ecount(random_ego_net)/((vcount(random_ego_net) * (vcount(random_ego_net) - 1))/2)
> ecount(random_ego_net)/((vcount(random_ego_net) * (vcount(random_ego_net) - 1))/2)
[1] 0.6Igraph有它自己的函数- graph.density,我们可以再次应用到数据中的每一个自我网络。
densities <- lapply(ego_nets, graph.density)
densities <- unlist(densities)
hist(densities)
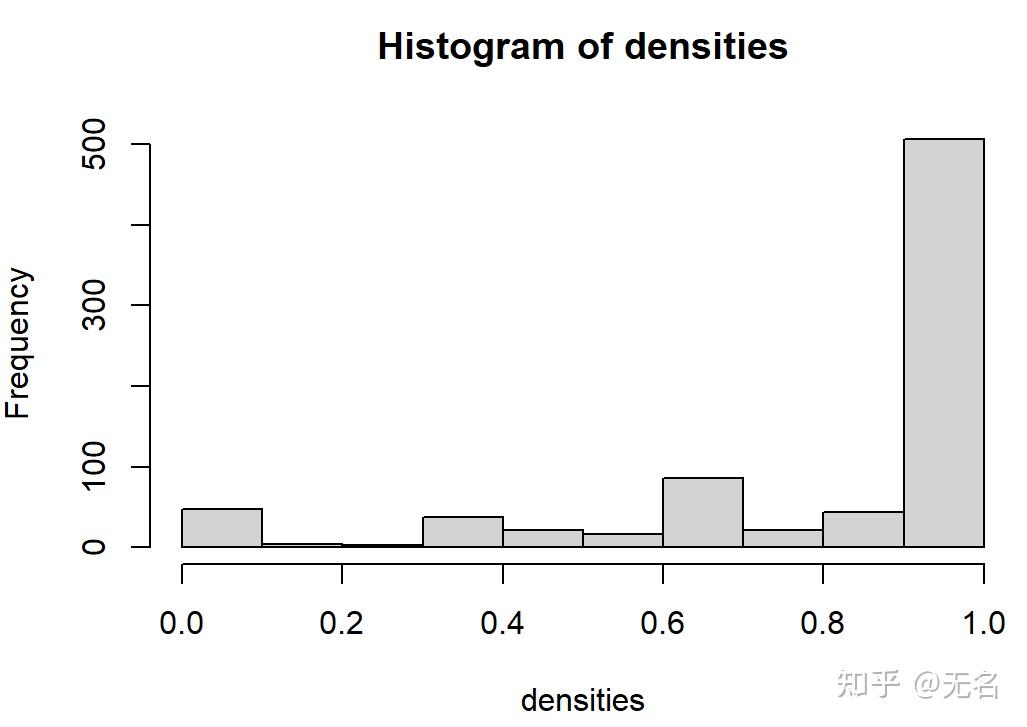
最后,我们画出l不同自我网络的密度分布。 |
|